お知らせ

- トップ
- お知らせ
RNA-分子間相互作用を大規模に解析する新たな技術を開発
-RNA標的低分子創薬への貢献に期待-
発表のポイント
● 数千のRNA(注1)構造に対する低分子の相互作用を一度の実験で解析できる技術を開発しました。
● 実施例として、本技術を用いて三種類のRNA結合性低分子の結合選択性(注2)を示しました。これまで評価が難しかったRNA構造に対する結合選択性評価を含め、本系で得られた情報が正しいことを実験的に確かめました。
● 本技術により得られたRNA結合選択性情報が、RNA結合分子探索法の一つである蛍光指示薬競合置換アッセイ(注3)のRNA標的を選択する際に有用であることを示しました。
概要
タンパク質を標的とした従来の創薬に代わり、RNAを標的とした低分子創薬の開発が、難治性疾患に対する新たな治療薬候補として注目されています。RNA結合分子の性質を理解して創薬に適した分子を設計するためには、対象分子のRNA結合選択性を大規模に調べる技術が非常に重要です。しかしこれまでの解析技術では、RNAに対する結合性情報を大規模に調査するのは困難でした。
東北大学多元物質科学研究所の鬼塚和光 准教授、永次 史 教授、大学院生の長澤瞭佑 氏(大学院理学研究科化学専攻)、京都大学iPS細胞研究所の齊藤博英 教授、小松リチャード馨 氏(当時 京都大学 大学院生)らは、マイクロRNA前駆体やウイルスRNAなどの数千種の部分構造に対する低分子のRNA結合選択性を大規模に解析する技術を開発しました。これまで結合選択性評価が難しかったRNA構造を含め、本系で得られた結合選択性情報が正しいことを実験的に確かめ、さらに得られた情報がRNA結合性分子の探索にも有用であることを示しました。
RNA結合性低分子の結合性の理解を深め、新たな分子の設計や探索にも役立つことから、RNA標的低分子創薬に大きく貢献すると期待できる成果です。
本研究成果は2024年5月1日、科学誌Communications Chemistryに掲載されました。
詳細な説明
研究の背景
低分子創薬における主な標的であったタンパク質の新規標的部位枯渇が近年問題視されるようになり、RNAを標的とした低分子創薬が注目されてきています。特に、メッセンジャーRNA(注4)、マイクロRNA前駆体(注5)のようなノンコーディングRNA(注6)、ウイルスや細菌のRNAが創薬標的として研究されています。しかしながら、標的RNAに選択的に作用する低分子は、多くの類似したRNA構造から標的のみを選択的に認識して強く結合する必要があるため、優れたRNA結合分子の開発は容易ではありません。そのため、標的特異的結合分子を創出する優れた評価法・探索法の開発が強く望まれています。
このような背景のもと、本研究グループは、RNA結合分子の選択性評価に着目しました。RNA結合分子の選択性を評価する場合、標的とする高次構造、その類似した構造に加え、コントロールとして一本鎖、二本鎖構造、異なる高次構造など10種類程度の配列に対する結合親和性を個別に評価し化合物の選択性を検証します。しかし、RNAは同じ種類の高次構造でも配列が変われば、その微細構造も変わるため、真の選択性を評価するためには、数百以上の網羅的な解析が不可欠であると考えられます。大規模解析において、次世代シーケンサー(注7)を利用した解析法が行われ始めていますが、G4重鎖構造(注8)などの複雑な構造では正確に選択性を評価できない問題がありました。
今回研究グループは、以前に開発されたRNA-タンパク質相互作用を大規模に解析するシステムであるFOREST(Folded RNA Element Profiling with Structure Library) (注9)を低分子解析用に改良することで、数千のRNA配列に対する低分子の結合親和性を一度のアッセイでランク化できる手法の構築を行いました。結果として得られる数千のRNAに対する親和性から、低分子の真の選択性の評価が可能になりました。
今回の取り組み
本解析には、調査する低分子を固定化したビーズ、RNAライブラリとDNAバーコードマイクロアレイ(注10)を用います(図1)。RNAライブラリ中のRNAは高次構造領域、共通ステム領域、バーコード領域からなり、3'末端を蛍光基で修飾したものを使います。高次構造領域にはマイクロRNA前駆体、リピートRNA、ウイルスRNAから部分構造を抽出した1824または3000種の構造が含まれています。このRNAライブラリから、ビーズに固定した分子に対して親和性の高いRNAを抽出します。この抽出したRNAはDNAバーコードマイクロアレイに付すことで、それぞれの固有のバーコード配列に従い、指定されたスポットへと集積します。ここでは、RNA バーコードが相補的な DNA バーコードとのみ二本鎖を形成することを利用しています。最後に、各スポットの蛍光強度を指標にRNA結合親和性のランクを決定することが可能になります。本法は数千から数万のRNA-分子間相互作用を一挙に解析することが原理上可能になります。
今回研究グループは、本技術を用いてG-clamp(注11)、チアゾールオレンジ-1(TO-1)、チアゾールオレンジ-3(TO-3)の三種類の低分子化合物のRNA結合選択性の評価を大規模に行いました(図2)。得られたRNA結合親和性情報を基に、RNAの一本鎖部と二本鎖部のA、G、C、Uの塩基数(注12)をランク間で比較することで、それぞれの化合物と結合親和性の高いRNA構造を考察しました。例えば、G-clampは一本鎖領域のグアニン塩基と結合親和性が高いこと、さらに変異実験(注13)の結果から特定のグアニン塩基を認識して強く結合していることが明らかになりました。また、大規模解析の結果に基づき、各親和性ランク帯から選んだ15~17種類のRNAに対するG-clampの結合力を蛍光滴定実験により調べたところ、大規模解析での結果と一致する結果が得られ、本解析情報が正しいことも実験的に確かめました(図3)。この際、これまで正確な評価が難しかったG4重鎖構造でも大規模解析と蛍光滴定実験の結果に相関があり、正しく評価できていることも確認できました。
さらにTO誘導体で得られたRNA結合親和性大規模情報は、市販の蛍光指示薬であるTO-PRO-1やTO-PRO-3を用いた蛍光滴定実験の結果とも強い相関を示しました。RNA結合分子探索法の一つである蛍光指示薬競合置換(FID)アッセイでは、蛍光指示薬が標的RNAと結合する必要があります。今回得られた蛍光指示薬の結合情報に基づき、標的RNA配列を選択し、化合物ライブラリを用いてFIDアッセイを行ったところ、結合親和性が中・上位であるRNA配列に対するアッセイの精度は高かった一方で、下位の配列では精度は低く、正確にRNA結合分子を探索することはできませんでした(図4)。この結果は、今回開発した大規模解析法がRNAと指示薬の適切な組み合わせを選ぶ際に有用であること、つまり得られた結合情報がFIDアッセイによるRNA結合分子探索にも役立つことを示しています。
今後の展開
今回開発した技術は、任意の低分子化合物に対するRNA結合親和性を大規模に解析することが可能になります。したがって、これまで見逃されていた標的の発見や真の選択性の理解に繋がることが期待できます。また得られたRNA選択性情報に基づき、安定なRNA―低分子複合体の組み合わせを選べるため、NMR(注14)やX線結晶構造解析に用いるRNA配列を選択する際にも役立つことが期待できます。これらの結合情報・構造情報を併せることで、今後さらに詳細なRNA結合性低分子の理解を進めることや、新たな分子の設計や探索にも役立つと考えられることから、本技術はRNA標的低分子創薬に大きく貢献することが期待できます。
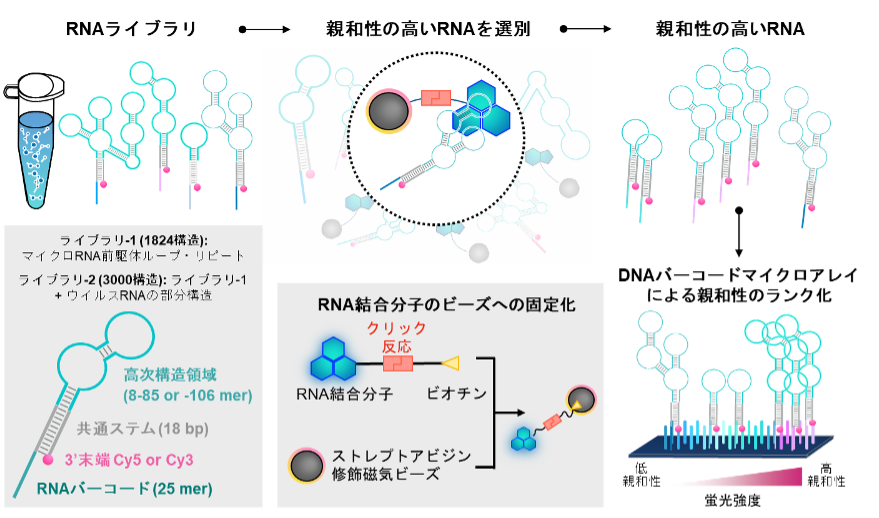
図1. 本技術によるRNA―分子間相互作用の大規模解析のスキーム図
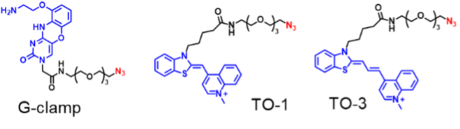
図2. 今回解析した分子の構造
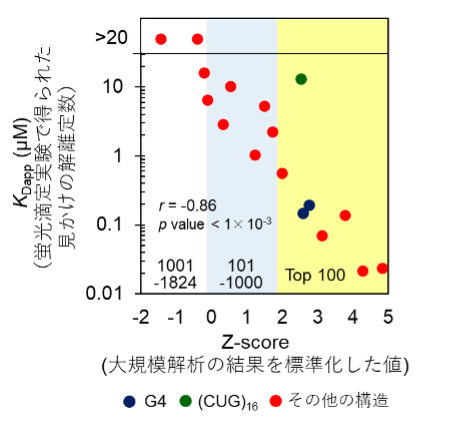
図3. 大規模解析によって得られたG-clampのRNA結合親和性と蛍光滴定実験で得られた見かけの解離定数との相関
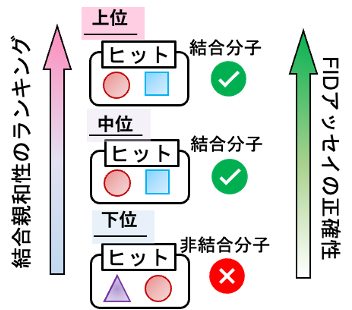
図4. FIDアッセイでのヒット化合物の正確性とRNA結合親和性のランキングの関係
謝辞
本研究は、JST創発的研究支援事業(JPMJFR2002)、次世代研究者挑戦的研究プログラム(JPMJSP2114)、JSPS科研費 基盤研究(B)(JP19H02845、JP20H02855、JP23H02076)、特別推進研究(JP20H05626)、新学術領域研究「化学コミュニケーションのフロンティア」(JP20H04762)、学術変革領域研究(A)「マテリアル・シンバイオシスのための生命物理化学」 (JP23H04051)、挑戦的研究(萌芽)(JP19K22387、JP21K19038)、公益財団法人 武田科学振興財団 研究助成、公益財団法人 上原記念生命科学財団 研究助成、公益財団法人野口研究所 野口遵研究助成、公益財団法人 東京生化学研究会 研究助成などの支援を受け実施されました。
用語説明
注1. RNA:リボ核酸の略。タンパク質をコードするメッセンジャーRNAだけでなく、コードしないノンコーディングRNAも存在し、その種類ごとに様々な機能を持つことが知られている。
注2.結合選択性:ここでは多数のRNAが存在する中で、低分子がどのような配列や構造を持つRNAに結合するのかを表した性質を意味する。
注3. 蛍光指示薬競合置換(FID)アッセイ:RNAと結合することで蛍光性が変化する蛍光指示薬を用いて、RNA結合分子を探索する手法。テスト化合物がRNAに結合するとき、蛍光指示薬を追い出すことで蛍光強度が変化するため、その蛍光強度の変化でヒット化合物を特定する。FIDアッセイでは、蛍光指示薬が結合可能な標的RNAを選択する必要があるが、その報告例は限られていた。
注4. メッセンジャーRNA:タンパク質をコードするRNA。DNAが持つ遺伝情報がタンパク質へと伝達される際の中間体である。
注5. マイクロRNA前駆体:ノンコーディングRNAの一つであるマイクロRNAの生合成過程の前駆体。マイクロRNAは癌や神経疾患などの様々な疾患に関与している。前駆体に強く結合し、生合成過程を阻害する分子は、それらの疾患に対して治療効果があると期待されている。
注6. ノンコーディングRNA:タンパク質をコードしないRNA。様々な機能を有し、種々の疾患に関与していることが解明されつつあり、特にマイクロRNAは創薬標的として注目されている。
注7. 次世代シーケンサー:多数のDNA配列を同時に特定できる技術。逆転写反応とポリメラーゼ連鎖反応(PCR)後に得られたDNAを配列解析することで、RNA結合分子の選択性評価に用いることが可能だが、複雑な高次構造を有する配列の正確な評価は難しいと考えられている。
注8. G4重鎖構造:4つのグアニンが形成するGカルテット平面からなる核酸高次構造。カリウムのようなカチオン存在下でこの平面構造が重なることで形成される。RNAでは翻訳やマイクロRNA生合成の調整など様々な働きを担っていると考えられている。
注9. FOREST(Folded RNA Element Profiling with Structure Library):詳しい技術の内容は下記を参照。
https://www.cira.kyoto-u.ac.jp/j/pressrelease/news/201209-090000.html
注10. DNAバーコードマイクロアレイ:DNAマイクロアレイは、スライドガラス上に多数の設計したDNAをスポット状に配置したものである。バーコードとはRNA構造ライブラリ中のRNA構造を識別するための固有の配列をここでは意味しており、DNAマイクロアレイ中のDNAはRNAライブラリ中の RNAが有するバーコード配列と相補的になるように今回設計した。これにより、RNAライブラリ中の各RNA構造を指定したスポットに配置し、定量する大規模解析が可能になる。
注11. G-clamp:4本の水素結合を介してグアニン塩基と強く結合する分子。グアニン塩基との結合様式がクランプのような形なのでG-clampと呼ばれている。元々は短い核酸の内部に組み込みグアニン塩基に対する認識能を上げる目的で使われていた。
注12. A、G、C、Uの塩基数:RNA中の塩基であるアデニン、グアニン、シトシン、ウラシルの塩基数。
注13. 変異実験:特定の塩基を変化させたRNAを用いて、元のRNAとの性質の違いを調べる実験。ここでは、一本鎖領域のグアニンをアデニンに変化させて、G-clampが結合するグアニン塩基の位置の特定を行った。
注14. NMR:強磁場を用いて分子の構造を特定する測定手法。RNAと低分子の複合体の構造を解析する際にも使われる。
論文情報
タイトル:Large-scale analysis of small molecule-RNA interactions using multiplexed RNA structure libraries
著者:Ryosuke Nagasawa*, Kazumitsu Onizuka*,**, Kaoru R. Komatsu*, Emi Miyashita, Hirotaka Murase, Kanna Ojima, Shunya Ishikawa, Mamiko Ozawa, Hirohide Saito**, and Fumi Nagatsugi**
*共同筆頭著者
**責任著者:東北大学多元物質科学研究所 准教授 鬼塚 和光
京都大学iPS細胞研究所(CiRA) 教授 齊藤 博英
東北大学多元物質科学研究所 教授 永次 史
掲載誌:Communications Chemistry
DOI:10.1038/s42004-024-01181-8
問い合わせ先
<研究に関すること>
東北大学多元物質科学研究所
准教授 鬼塚 和光
TEL: 022-217-5634
Email: onizuka[at]tohoku.ac.jp
東北大学多元物質科学研究所
教授 永次 史
TEL: 022-217-5633
Email: nagatugi[at]tohoku.ac.jp
(報道に関すること)
東北大学多元物質科学研究所 広報情報室
TEL: 022-217-5198
Email: press.tagen[at]grp.tohoku.ac.jp
京都大学iPS細胞研究所(CiRA)国際広報室
https://www.cira.kyoto-u.ac.jp/j/contact/
*[at]を@に置き換えてください
Posted on:2024年5月10日